首页 >> 生活 >
开发从单细胞转录组数据推断基因调控网络的新方法
单细胞RNA测序(scRNA-seq)技术提供了了解单细胞分辨率调控机制的机会。基因调控网络(GRN)为细胞系统中的调控机制提供了重要的蓝图,因此在生物学研究中发挥着核心作用。因此,必须开发一种准确的工具,从scRNA-seq数据中推断GRN。
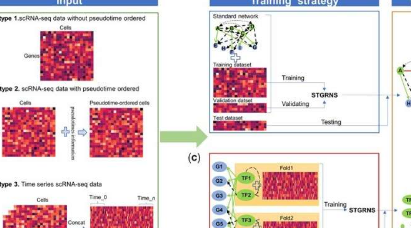
中国科学院武汉植物园的研究人员开发了一种新方法,即STGRNS,用于使用深度学习模型从scRNA-seq数据构建GRN。结果已发表在《生物信息学》上,工具和教程可在https://github.com/zhanglab-wbgcas/STGRNS公开获得。
在该算法中,提出了一种基因表达基序技术,将每个基因对转换为可以作为变压器编码器接收的形式。通过避免网络中缺少阶段特异性调控,STGRNS可以从静态、伪时间或时间序列单细胞转录组数据中准确推断GRN。
研究人员表明,STGRNS在48个基准数据集上优于其他最先进的深度学习方法,包括21个静态scRNA-seq数据集和27个时间序列scRNA-seq数据集。
与其他基于“黑匣子”深度学习的方法不同,这些方法通常以不透明和为预测提供明确理由的相关困难为特征,STGRNS更可靠并且可以解释预测。
此外,与其他基于深度学习模型的GRN重构方法相比,STGRNS具有较少的超参数,这是其泛化性优异的主要原因之一。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
分享:
最新文章
-
宝子们,杭州 房子装修完成啦!这次要给大家分享几家设计超赞的装修公司哦。它们各具特色,从空间规划到风格...浏览全文>>
-
欲筑室者,先治其基。在上海,装修房子对于每个业主而言,都是极为关键的一步,然而,如何挑选一家值得信赖的...浏览全文>>
-
2025年以来,联通支付严格贯彻落实国家战略部署,以数字和科技为驱动,做好金融五篇大文章,履行支付为民社会...浏览全文>>
-
良工巧匠,方能筑就华居;精雕细琢,方可打造美家。当我们谈论装修公司时,选择一家靠谱可靠的公司是至关重要...浏览全文>>
-
在当今社会,随着城市化进程的高速推进,建筑垃圾的产生量与日俱增。据权威数据显示,我国每年建筑垃圾产生量超 ...浏览全文>>
-
家人们,在上海要装修,选对公司那可太重要了!古语有云:"安得广厦千万间,大庇天下寒士俱欢颜。"一个温馨的...浏览全文>>
-
近年来,新能源汽车市场发展迅猛,各大品牌纷纷推出各具特色的车型以满足消费者多样化的需求。作为国内新能源...浏览全文>>
-
近年来,随着汽车市场的不断变化和消费者需求的升级,安徽滁州地区的宝来2025新款车型在市场上引起了广泛关注...浏览全文>>
-
随着汽车市场的不断变化,滁州地区的消费者对高尔夫车型的关注度持续上升。作为大众品牌旗下的经典车型,高尔...浏览全文>>
-
在2023年,大众探影以其时尚的设计和出色的性能赢得了众多消费者的青睐。作为一款小型SUV,探影凭借其紧凑的车...浏览全文>>
大家爱看
频道推荐
站长推荐
- 安徽滁州途安L新车报价2022款,最低售价16.68万起,入手正当时
- 小鹏G7试驾,新手必知的详细步骤
- 别克GL8预约试驾,4S店的贴心服务与流程
- 安徽阜阳ID.4 CROZZ落地价全解,买车必看的省钱秘籍
- 淮北探岳多少钱 2025款落地价,最低售价17.69万起现在该入手吗?
- 安徽淮南大众CC新款价格2025款多少钱能落地?
- 淮北长安启源C798价格,最低售价12.98万起现在该入手吗?
- 安徽淮南途锐价格,各配置车型售价全解析
- 蒙迪欧试驾预约,4S店体验全攻略
- 沃尔沃XC40试驾需要注意什么
- 滁州ID.4 X新车报价2025款,各车型售价大公开,性价比爆棚
- 试驾思域,快速操作,轻松体验驾驶乐趣
- 试驾长安CS35PLUS,一键搞定,开启豪华驾驶之旅
- 天津滨海ID.6 X落地价限时特惠,最低售价25.9888万起,错过不再有
- 天津滨海凌渡多少钱?看完这篇购车攻略再做决定
- 安徽池州长安猎手K50落地价,买车前的全方位指南
- 山东济南ID.6 CROZZ 2024新款价格,最低售价19.59万起,现车充足
- 试驾海狮05EV,新手必知的详细步骤
- 生活家PHEV多少钱 2025款落地价走势,近一个月最低售价63.98万起,性价比凸显
- 奇瑞风云A9试驾,新手必知的详细步骤
